Código
library(tidyverse)
library(pdftools)
library(rvest)
library(httr)
library(showtext)
library(ggtext)O Congresso Brasileiro de Ciência do Solo (CBCS) é um evento técnico-científico que ocorre a cada dois anos (anos ímpares). O objetivo deste evento é reunir alunos, professores, pesquisadores e profissionais das áreas afins para a troca de conhecimento e discussão sobre as futuras perspectivas da ciência do solo.
Esse ano o XXXVIII CBCS ocorreu em Florianopolis – SC, concomitante a ele ocorreu o XXIII Congresso Latino-Americado de Ciência do solo (CLACS). O evento em conjunto foi chamado de SOLOS FLORIPA 2023, com organização das sociedades Latino-americana (SLCS) e Brasileira (SBCS) da Ciência do Solo e realização da Empresa de Pesquisa Agropecuária e Extensão Rural de Santa Catarina (Epagri).
Uma das características mais importante do congresso é a possibilidade de os “pesquisadores” apresentarem trabalhos técnico-científicos sobre seus respectivos objetos de estudo. Diante disso, surge a dúvida em saber quais as áreas/subáreas teve as maiores quantidades de trabalhos apresentados e talvez entender o foco das pesquisas em Ciência do Solo na atualidade. Para sanar essa dúvida (pelo menos parcialmente) apresento nesse post uma forma de retirar os dados do site do congresso e realizar uma apresentação gráfica sobre os trabalhos. Para isso utilizo a linguagem de programação R.
Para mais informações acesse o site: Site fora do ar
Para essa análise vou utilizar os seguintes pacotes do R
Atualmente, a SBCS possui 4 divisões científicas com 16 subdivisões, sendo:
Os dados do número de trabalhos está na página de Trabalho Aprovados no site. A captura dos dados foi realizada com o endereço eletrônico da página. Um avaliação prévia de como se conportava a página e como os dados aparaciam foi realizada.
Um loop foi realizado para remover os dados de cada uma das áreas e subareas.
df <- as.data.frame(x = 1:length(id))
for (i in 1:16) {
dados <- httr::GET(paste0(primeira_parte, id[i], segunda_parte))
get_content <- httr::content(dados)
raw_table <- get_content |>
rvest::html_nodes(".container-fluid")
df[i, ] <-
raw_table[[3]] |>
rvest::html_text() |>
strsplit("\n") |>
unlist() |>
enframe(name = NULL, value = "linha") |>
dplyr::filter(stringr::str_detect(linha, pattern = ".*registro"))
}Além de captar as iformações, foi realizado um processamento e manipulação para organizar os dados.
divi <- raw_table[[3]] |>
rvest::html_text() |>
strsplit("\n") |>
unlist() |>
enframe(name = NULL, value = "linha") |>
dplyr::filter(stringr::str_detect(linha, pattern = " TODASDivisã")) |>
str_split(pattern = "Di", simplify = T) |>
as.data.frame() |>
pivot_longer(cols = 2:17, names_to = "Divi", values_to = "nome") |>
filter(Divi != "V1") |>
mutate(di = "Di") |>
transmute(Divi = paste(di, nome, sep = ""))
d_final <- cbind(divi, df)
dados.ok <- d_final |>
mutate(
partici = str_extract(`1:length(id)`,
pattern = "[:digit:]{1,}"
),
partici = as.numeric(partici)
)
comi <- str_split(d_final$Divi, ":")
data_separado <- data.frame(do.call(rbind, comi))
df_ok <- cbind(data_separado, dados.ok)
df_ok <- df_ok |> mutate(remover = str_sub(df_ok$X2, start = 16, end = 90))
font_add("Didot", "GFS_Didot/GFSDidot-Regular.ttf")
showtext_auto()Com os dados devidamente organizados foi realizado a visualização gráfica.
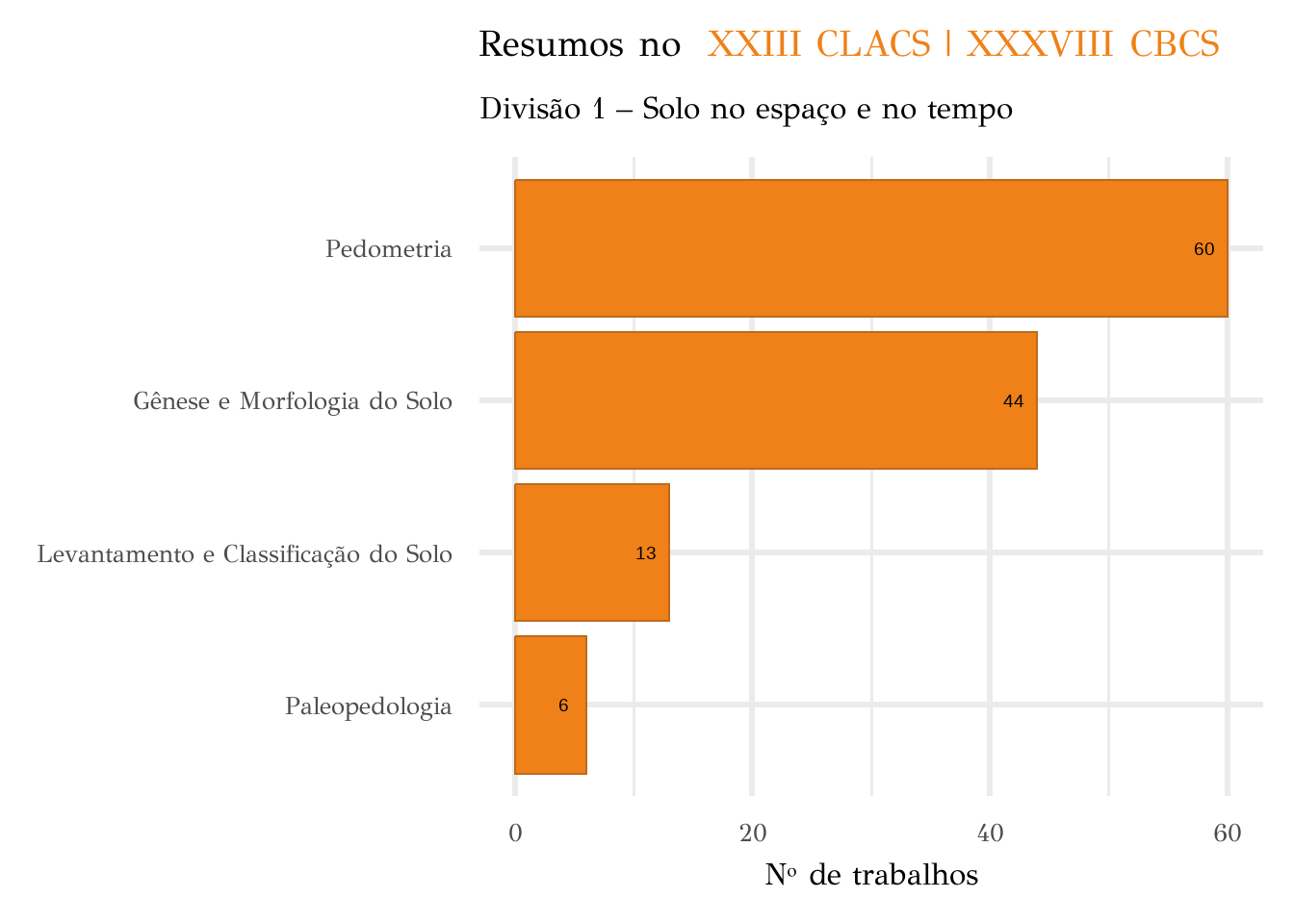
df_ok |>
filter(X1 == "Divisão 1 – Solo no espaço e no tempo") |>
ggplot(aes(y = partici, x = fct_reorder(remover, partici))) +
coord_flip() +
geom_col(fill = "#ef8118", col = "#bc6a1d") +
# facet_wrap(~X1, scales = "free") +
labs(
x = NULL, y = "Nº de trabalhos",
title = 'Resumos no <span style = "color:#ef8118"> XXIII CLACS | XXXVIII CBCS </span>',
subtitle = "Divisão 1 – Solo no espaço e no tempo"
) +
geom_text(aes(label = partici), nudge_y = -2, col = "black", size = 5) +
theme_minimal(24) +
theme(
text = element_text(family = "Didot"),
plot.title = ggtext::element_markdown()
)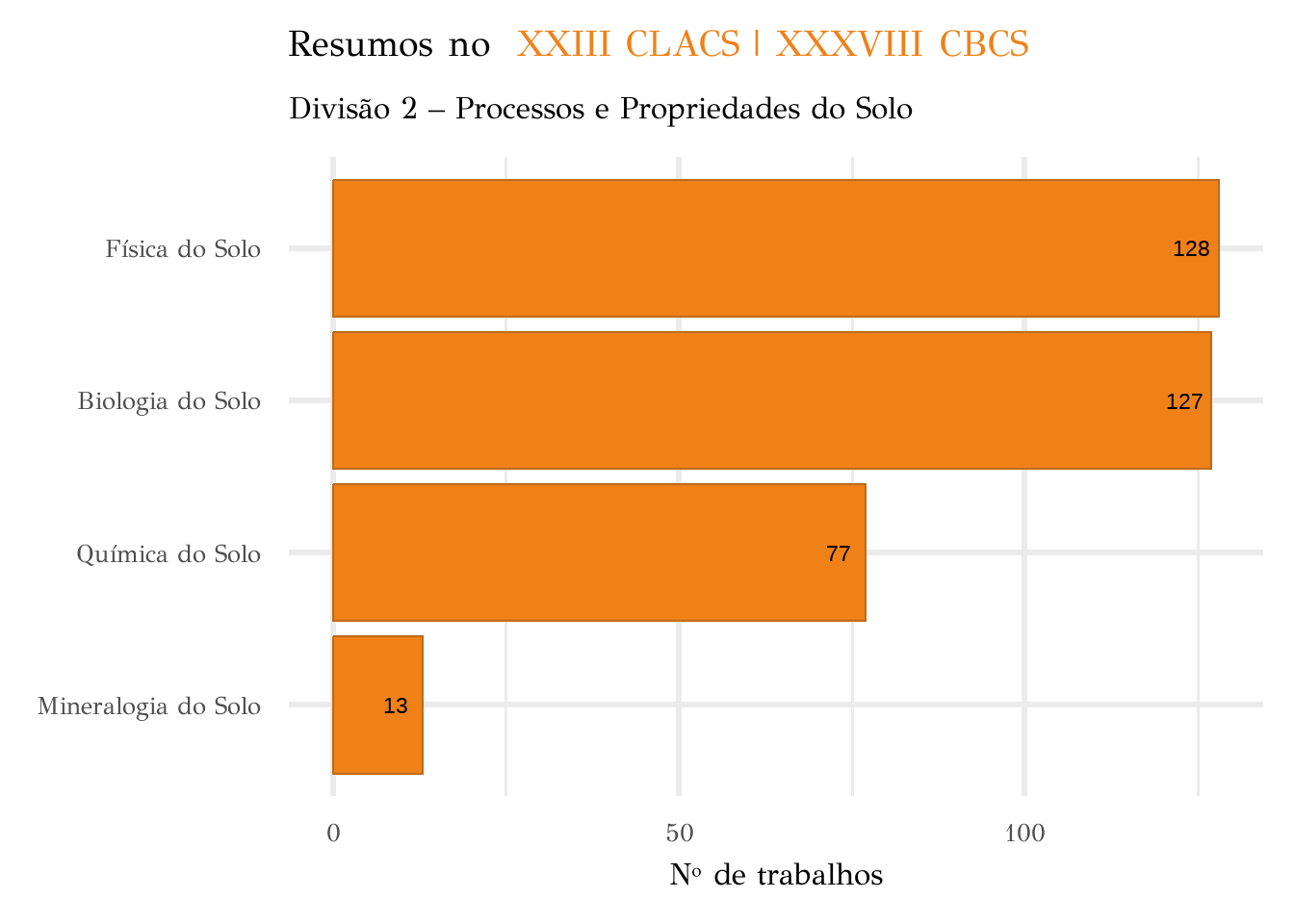
df_ok |>
filter(X1 == "Divisão 2 – Processos e Propriedades do Solo") |>
ggplot(aes(y = partici, x = fct_reorder(remover, partici))) +
coord_flip() +
geom_col(fill = "#ef8118", col = "#bc6a1d") +
# facet_wrap(~X1, scales = "free") +
labs(
x = NULL, y = "Nº de trabalhos",
title = 'Resumos no <span style = "color:#ef8118"> XXIII CLACS | XXXVIII CBCS </span>',
subtitle = "Divisão 2 – Processos e Propriedades do Solo"
) +
geom_text(aes(label = partici), nudge_y = -4, col = "black", size = 6) +
theme_minimal(24) +
theme(
text = element_text(family = "Didot"),
plot.title = element_markdown()
)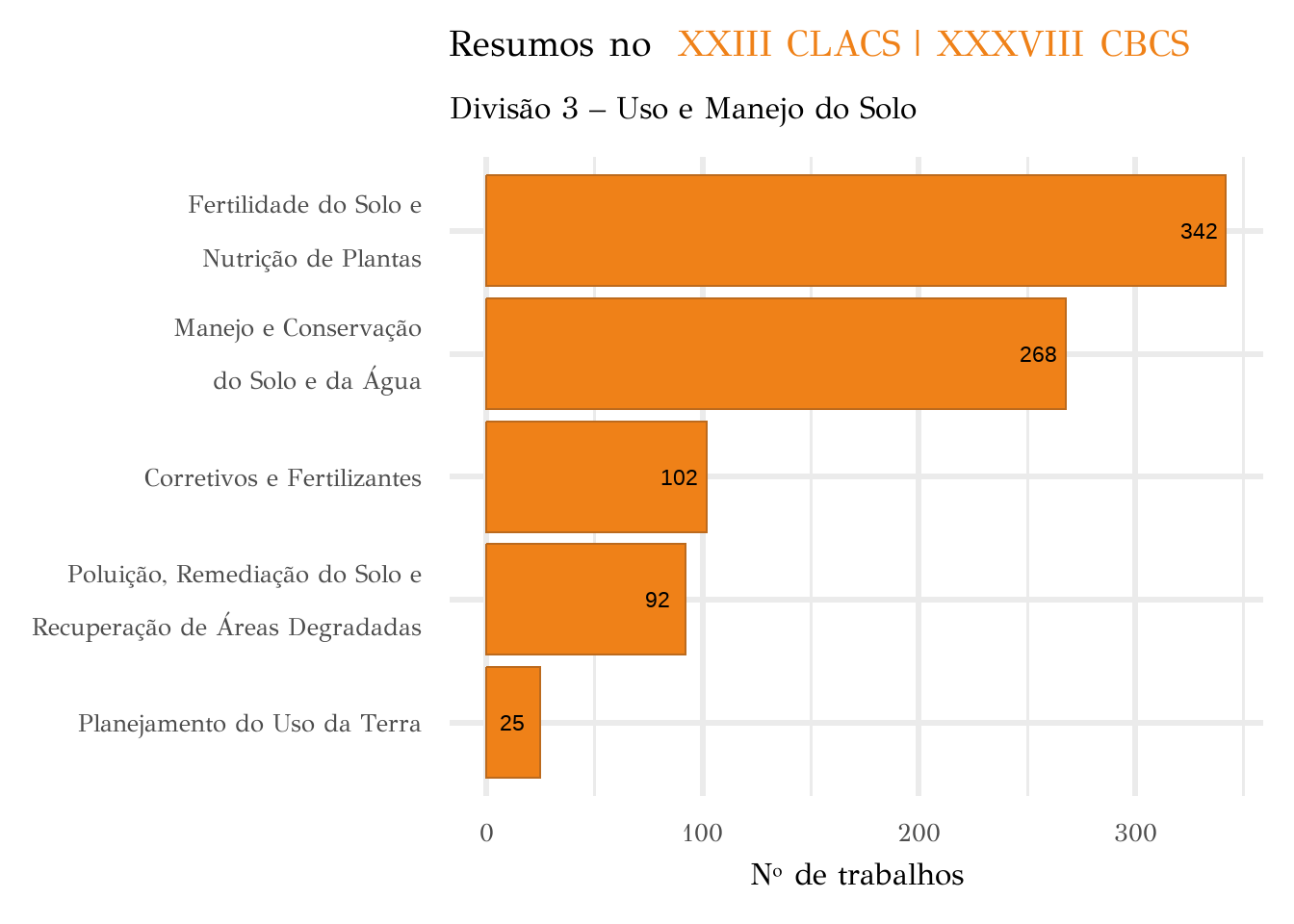
df_ok |>
filter(X1 == "Divisão 3 – Uso e Manejo do Solo") |>
mutate(remover = factor(remover, levels = c(
" Fertilidade do Solo e Nutrição de Plantas",
" Corretivos e Fertilizantes",
" Manejo e Conservação do Solo e da Água",
" Planejamento do Uso da Terra",
" Poluição, Remediação do Solo e Recuperação de Áreas Degradadas"
), labels = c(
"Fertilidade do Solo e \n Nutrição de Plantas",
"Corretivos e Fertilizantes",
"Manejo e Conservação \n do Solo e da Água",
"Planejamento do Uso da Terra",
"Poluição, Remediação do Solo e \n Recuperação de Áreas Degradadas"
))) |>
ggplot(aes(y = partici, x = fct_reorder(remover, partici))) +
coord_flip() +
geom_col(fill = "#ef8118", col = "#bc6a1d") +
# facet_wrap(~X1, scales = "free") +
labs(
x = NULL, y = "Nº de trabalhos",
title = 'Resumos no <span style = "color:#ef8118"> XXIII CLACS | XXXVIII CBCS </span>',
subtitle = "Divisão 3 – Uso e Manejo do Solo"
) +
geom_text(aes(label = partici), nudge_y = -13, col = "black", size = 6) +
theme_minimal(24) +
theme(
text = element_text(family = "Didot"),
plot.title = element_markdown()
)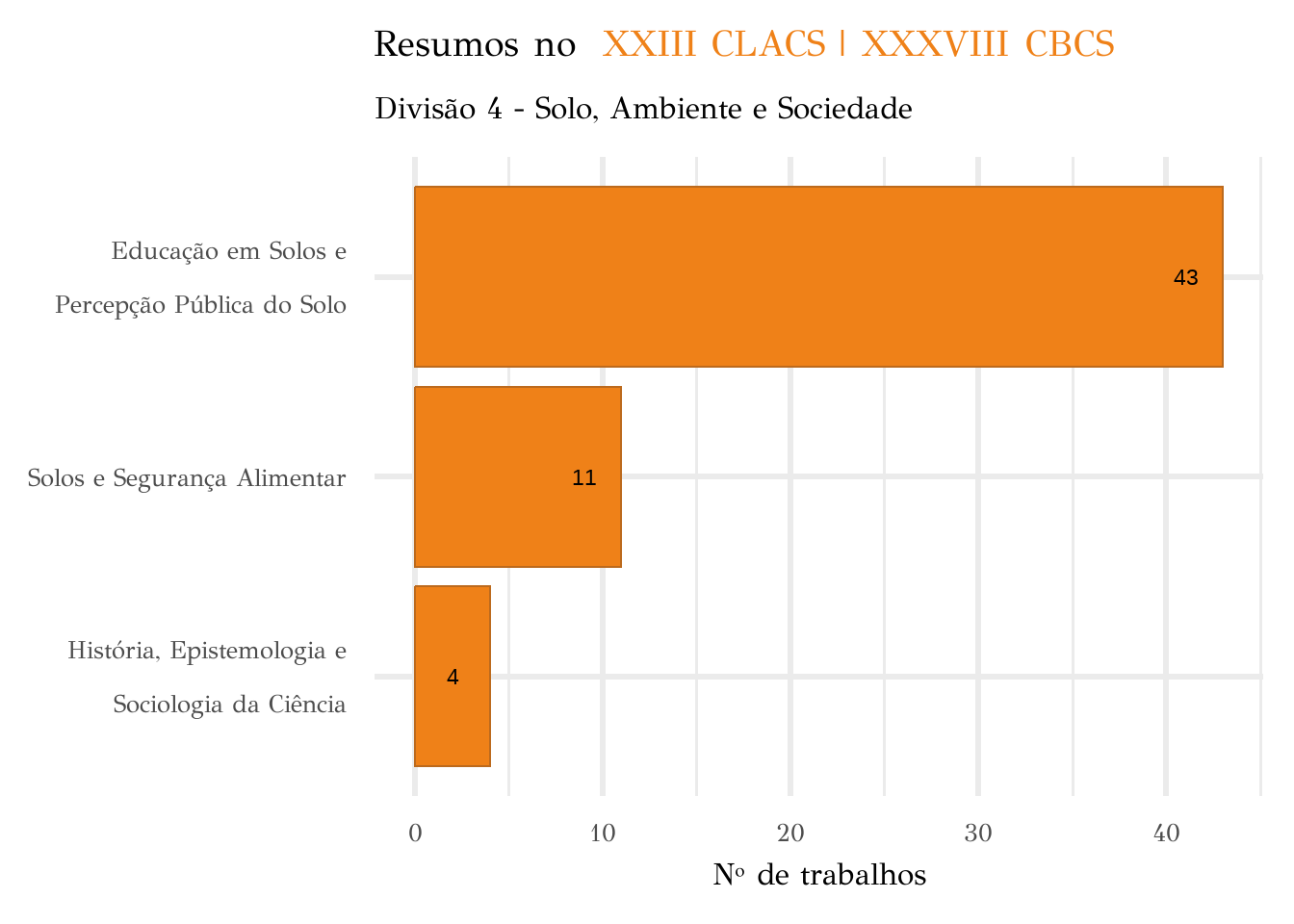
df_ok |>
filter(X1 == "Divisão 4 - Solo, Ambiente e Sociedade") |>
mutate(remover = factor(remover, labels = c(
"Educação em Solos e \n Percepção Pública do Solo",
"História, Epistemologia e \n Sociologia da Ciência",
"Solos e Segurança Alimentar"
))) |>
ggplot(aes(y = partici, x = fct_reorder(remover, partici))) +
coord_flip() +
geom_col(fill = "#ef8118", col = "#bc6a1d") +
# facet_wrap(~X1, scales = "free") +
labs(
x = NULL, y = "Nº de trabalhos",
title = 'Resumos no <span style = "color:#ef8118"> XXIII CLACS | XXXVIII CBCS </span>',
subtitle = "Divisão 4 - Solo, Ambiente e Sociedade"
) +
geom_text(aes(label = partici), nudge_y = -2, col = "black", size = 6) +
theme_minimal(24) +
theme(
text = element_text(family = "Didot"),
plot.title = element_markdown()
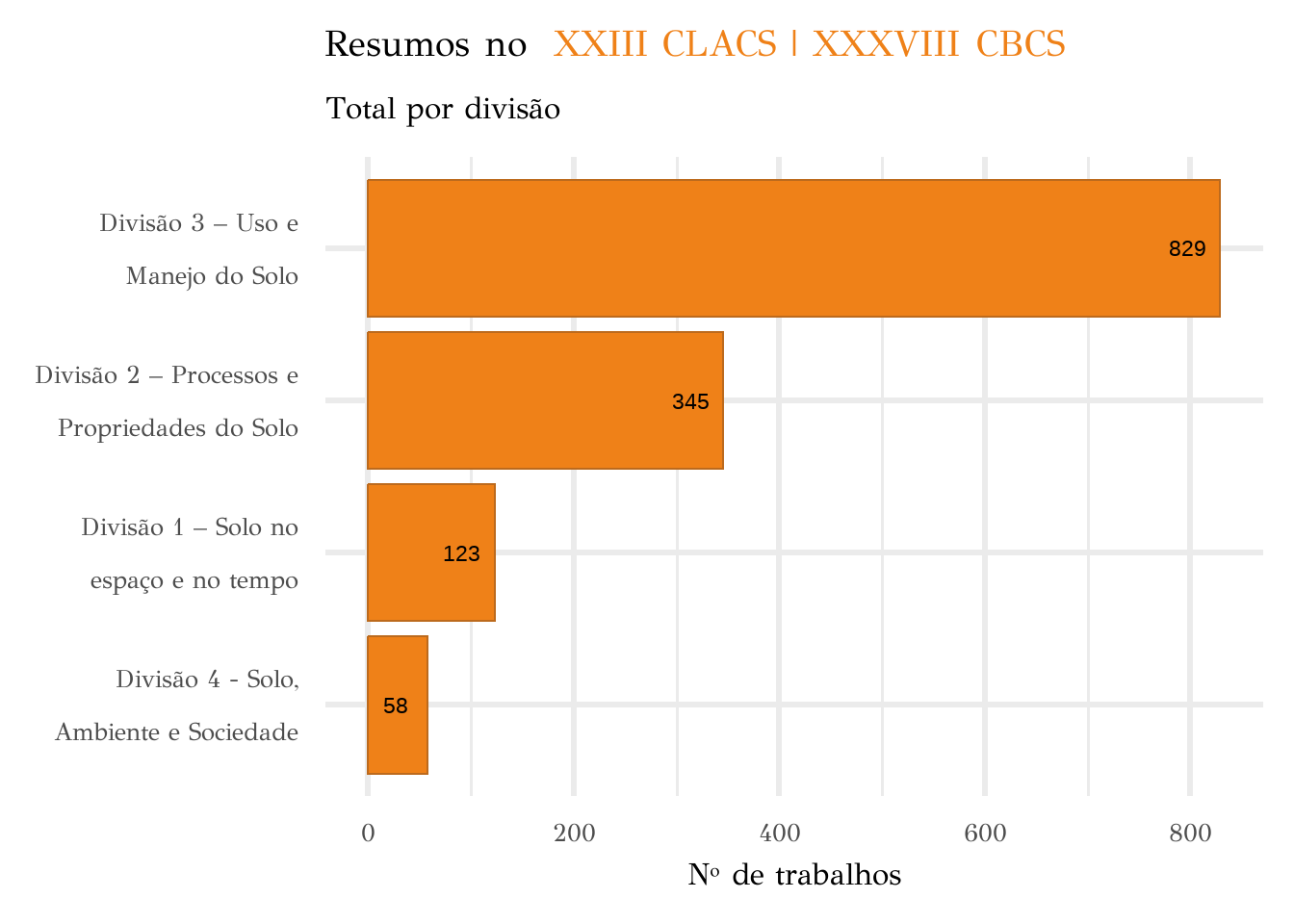
)De forma geral, observa-se que a maioria dos trabalhos submetidos e apresentados no evento pertence à divisão científica 3 – Uso e Manejo do Solo, totalizando 829 trabalhos. Em contrapartida, a divisão 4 – Solo, Ambiente e Sociedade apresentou o menor número de trabalhos, com apenas 58.
df_ok |>
group_by(X1) |>
summarise(soma = sum(partici)) |>
mutate(
X1 = factor(X1, labels = c(
"Divisão 1 – Solo no \n espaço e no tempo",
"Divisão 2 – Processos e \n Propriedades do Solo",
"Divisão 3 – Uso e \n Manejo do Solo",
"Divisão 4 - Solo, \n Ambiente e Sociedade"
)),
total = sum(soma)
) |>
ggplot(aes(y = soma, x = fct_reorder(X1, soma))) +
coord_flip() +
geom_col(fill = "#ef8118", col = "#bc6a1d") +
# facet_wrap(~X1, scales = "free") +
labs(
x = NULL, y = "Nº de trabalhos",
title = 'Resumos no <span style = "color:#ef8118"> XXIII CLACS | XXXVIII CBCS </span>',
subtitle = "Total por divisão"
) +
geom_text(aes(label = soma), nudge_y = -32, col = "black", size = 6) +
theme_minimal(24) +
theme(
text = element_text(family = "Didot"),
plot.title = element_markdown()
)Os códigos apresentados foram produzidos sem critérios de qualidade. Melhorias ainda podem ser feitas.